鉴于该病毒对人类的潜在威胁以及S蛋白在冠状病毒入侵过程中的决定性作用,2020年8月20日,福建师范大学欧阳松应教授课题组联合中科院生物物理研究所朱平研究员课题组在Journal of Virology上在线发表题为“Cryo-electron microscopy structure of the SADS-CoV spike glycoproteinprovides insights into an evolution of unique coronavirus spike proteins”的研究论文,研究人员通过Cryo-EM解析了S蛋白3.55 Å的结构,并从结构层面系统的分析比较了冠状病毒S蛋白整体结构、N端结构域(NTD)和C端结构域(CTD)的结构及其进化谱系。

冠状病毒S蛋白都以三聚体形态存在,依据其亚基的排列模式可以分为两类:交叉排列型(cross-subunit mode)和内嵌排列型(intra-subunit mode),其中前者的代表病毒为:β冠状病毒和γ冠状病毒,后者的代表病毒为α冠状病毒和δ冠状病毒,SARS-CoV 的S蛋白就属于典型的内嵌型排列模式。尽管α冠状病毒都采用内嵌型排列模式,但是在病毒的进化过程中,由于其NTD的类型和个数的不同,它又可以大概分为三类:只拥有1个NTD的α冠状病毒,这个NTD可以是一类NTD(subtype I NTD,例如:SADS-CoV),也可以是二类NTD (subtype II NTD,例如:HCoV-229E);拥有两个NTD的α冠状病毒,其中I类和II类NTD各一个(例如:HCoV-NL63,PEDV和FCoV)。因此,冠状病毒的NTD在进化过程,很可能以两种不同类型的α冠状病毒NTD做为祖先,分别进化的同时,也在某一时刻因发生重组而演变出具有两个NTD的α冠状病毒。其中一类NTD分别独立的进化为β和γ冠状病毒的NTD,二类NTD则独立进化为δ冠状病毒的NTD。对于CTD来说,依据其核心区β折叠层的数目可以分为两类: one-layerCTD和two-layer CTD。其中SADS-CoV属于前者,而其它α冠状病毒的CTD则属于后者。
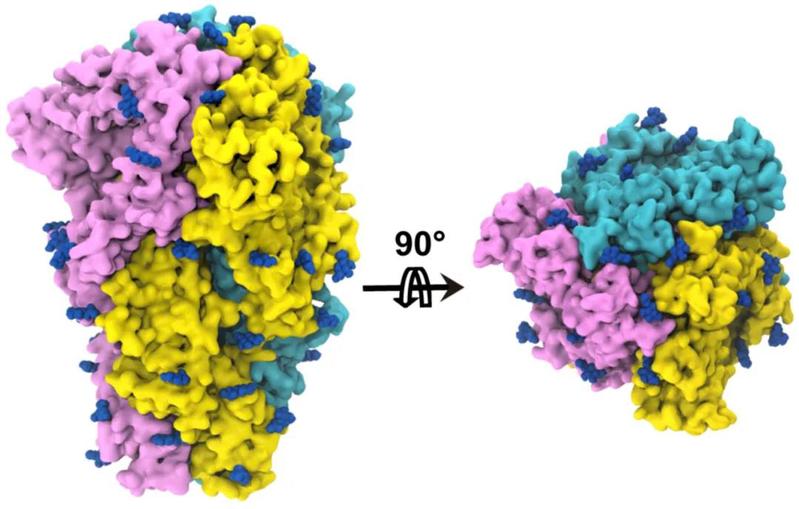
在进化关系上,α冠状病毒的one-layer CTD可能在进化过程中重组了类似于β冠状病毒受体结合区(insertion domain)样结构并最终进化成β冠状病毒的CTD;其它α冠状病毒和δ冠状病毒的two-layerCTD经过演变最终进化为γ冠状病毒的CTD。综合NTD、CTD以及整个S蛋白的结构特征,文章认为SADS-CoV很可能也采用了经典的双受体结合识别系统,即NTD作为糖基受体,而CTD作为蛋白受体,共同完成病毒与宿主的识别过程。同时作者基于结构的比较分析,推测SADS-CoV利用其CTD作为受体识别区,并发现该病毒在逃避宿主免疫识别方面的几个特点,比如其S蛋白各亚基的堆积更加紧凑,同一亚基的NTD与CTD存在相互作用,CTD受体结合区域被一个糖基遮蔽等,这些结构特点的分析将为基于S蛋白的疫苗的研发提供有力的支持。
据悉,该工作由福建师范大学生命科学学院、南方生物医学研究中心欧阳松应教授和中科院生物物理所朱平研究员合作完成,欧阳松应课题组关洪鑫副研究员和生物物理所博士生王有望为本文共同第一作者。