CRISPR-Cas系统是原核生物中形成一种能够抵御噬菌体和外来核酸入侵的防御系统,该系统可以对生物体基因组或核酸物质进行序列特异性的删除、替换或者突变,从而实现人为改变或干预生物体的遗传物质,尤其是CRISPR-Cas系统第二大类的成员(比如CRISPR-Cas9系统)在生物学及医学领域具有巨大的应用价值。
2021年6月9日,《Nature》子刊《Nature Communications》在线发表了福建师范大学欧阳松应教授课题组关于CRISPR-Cas系统第二大类效应蛋白成员Cas12i1的最新研究成果,论文标题为“Mechanistic insights into the R-loop formation and cleavage in CRISPR-Cas12i1”。该研究报道了毛螺菌科细菌(Lachnospiraceae bacterium ND2006)的Cas12i1效应蛋白与CRISPR RNA(简称crRNA)及双链靶DNA形成的三元复合物两种状态的晶体结构,其中一种状态为Cas12i1、crRNA及双链靶DNA形成所谓“R-loop”的三元复合物结构,另一种状态为R-loop结构中的双链靶DNA被Cas12i1切割后的三元复合物结构;该研究通过结构分析和生化实验,鉴定并验证了Cas12i1参与双链靶DNA的识别、解旋及切割的关键蛋白基序及氨基酸残基,以及决定具体PAM序列的关键氨基酸残基。此外,该研究还报道了Cas12i1与crRNA形成的二元复合物的晶体结构;通过比较Cas12i1二元及三元复合物之间的构象变化,较为完整地揭示了双链靶DNA结合从而激活Cas12i1的DNase活性的分子机理。该研究结果为将来在基因编辑工作中理性使用CRISPR-Cas12i1系统提供了结构基础及分子机理的指导。这是该课题组继2018年CRISPR-Cas13b(详见BioArt报道:Cell Research | 欧阳松应组在CRISPR-Cas13b结构与功能方面取得重要进展)、2019年CRISPR-Cas13d(详见BioArt报道:Nat Comm | 欧阳松应课题组发表CRISPR-Cas13d最新研究成果)的结构与功能研究之后的又一研究成果。最近,该课题组又在此两项工作基础上对CRISPR-Cas13系统效应蛋白的结构与功能进行了总结(详见BioArt报道:Adv Sci综述 | 欧阳松应团队总结细菌CRISPR-Cas13系统效应蛋白结构与功能机制、应用及研究前景)。

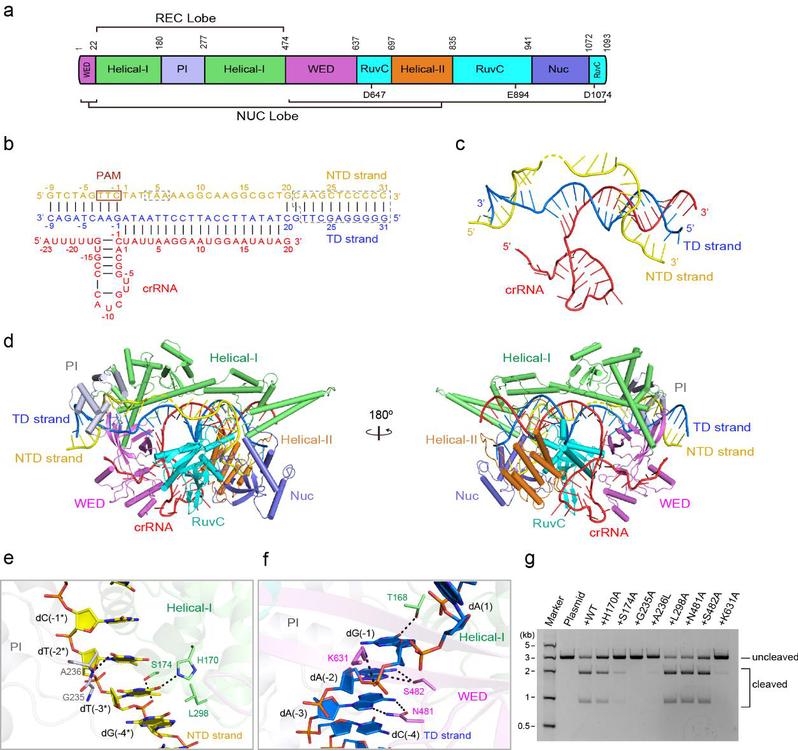
图1. Cas12i1的pre-cleavage R-loop三元复合物的晶体结构
2019年1月,美国Arbor生物技术公司鉴定出与已知第二大类Cas效应蛋白存在明显差异的多个Cas效应蛋白。其中,Cas12i在crRNA的引导下可以切割双链DNA底物,另外还发现Cas12i切割双链靶DNA的非靶标DNA链(non-target DNA strand,NTD strand)比靶标DNA链(target DNA strand,TD strand)效率更高;由于Cas12i的分子量比已经进入工程应用的Cas9、Cas12a及Cas12b的分子量相对偏小,这有利于它被包装到容量有限的应用载体中发挥作用,然而它切割DNA两条链的分子机理以及切割效率为何明显不同尚不清楚。针对上述问题,关于CRISPR-Cas12i1和CRISPR-Cas12i2的两项结构生物学研究成果分别于2020年9月和10月在线发表(详见BioArt报道:NSMB | 畅磊福团队阐明CRISPR-Cas12i系统的工作机理;Nat Comm | 王艳丽组和龚为民组合作揭示Cas12i2 双镁离子依赖的DNA切割机制)。而本项研究报道的Cas12i1效应蛋白的三种晶体结构提供了额外且新颖的信息。首先,本项研究以2.75Å和2.45Å的分辨率分别报道了双链靶DNA被Cas12i1切割前(pre-cleavage)及切割后(post-cleavage)的两个R-loop三元复合物结构信息;在这两个R-loop复合物结构中,双链靶DNA中的靶标DNA链与非靶标DNA链解旋,然后靶标DNA链与crRNA的向导序列(guide region)结合并形成19-bp的DNA-RNA异源双链结构(heteroduplex);这与之前报道的Cas12i1和Cas12i2分别结合28-bp和26-bp的DNA-RNA异源双链结构有所不同,这意味着完整的R-loop结构可以影响靶标DNA链与crRNA向导序列形成的异源双链的长度,而本项研究观察到了较为完整的R-loop结构(图1)。然后,一个被称为“拉链基序(zipper motif)”的关键蛋白基序(位于其Helical-I结构域的氨基酸残基160-177aa)和两对“磷酸基团锁”(phosphate lock)(氨基酸残基R12与K483、R535与K923)被鉴定出来,它们可以促进双链靶DNA在紧邻PAM序列下游解旋,以便于靶标DNA链与crRNA的向导序列形成异源双链结构(图2)。另外,研究还发现Cas12i1的PI结构域的G235和A236残基在决定具体的PAM序列时扮演关键作用(图1)。
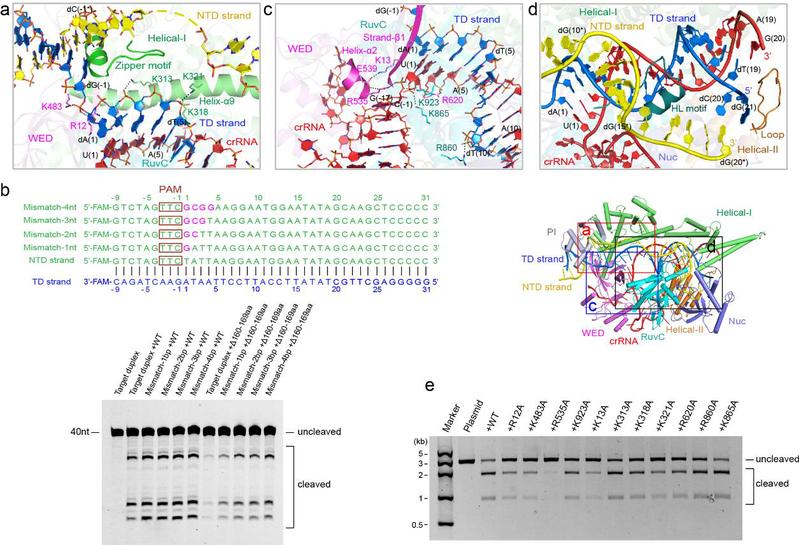
图2. Cas12i1结合的双链靶DNA的解旋与R-loop结构的形成
进一步的体外酶切实验表明,Cas12i1首先在PAM序列下游第13-15个核苷酸位置处连续切割非靶标DNA链,随后在PAM序列下游第24个核苷酸位置处切割靶标DNA链(图3)。在post-cleavage R-loop三元复合物结构中,双链靶DNA中的非靶标DNA链与靶标DNA链已经被Cas12i1酶切,其中有趣的是切割后的非靶标DNA链(non-target DNA strand)的一端从该三元复合物延伸出来,并被晶体内与之相邻的另一个三元复合物的RuvC结构域DNase活性中心重新捕获,从而进一步提供了Cas12i1反式切割(trans-cleavage)DNA底物的结构信息(图3)。此外,在pre-cleavage R-loop三元复合物结构中,位于其Helical-II结构域的一个loop区(氨基酸残基724-737aa)可以帮助靶标DNA链的5'-末端转向非靶标DNA链的3'-末端(图2),而且该loop区还有助于将靶标DNA链加载到RuvC结构域的DNase活性位点,从而使靶标DNA链被切割。
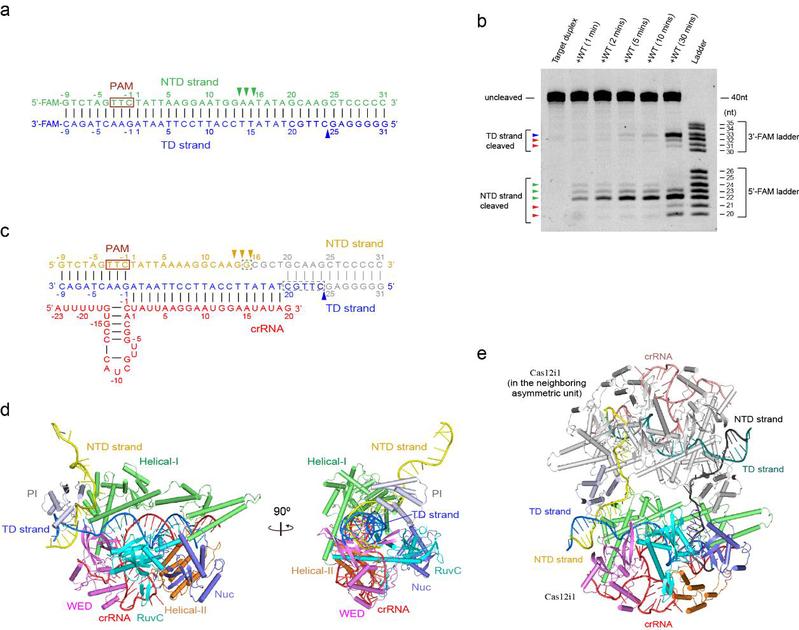
图3. Cas12i1的post-cleavage R-loop三元复合物的晶体结构
在Cas12i1的二元复合物结构中,研究发现位于crRNA向导序列的种子区域(seed region)互补结合了一段来自宿主细胞的3-nt伪靶标核酸(pseudo-target),这些在crRNA种子区域内的碱基配对在一定程度上模拟了Cas12i1初始识别与结合靶核酸时的场景(图4)。进一步比较Cas12i1的二元及三元复合物的构象变化,可以发现Cas12i1与双链靶DNA结合后,PI结构域发生了剧烈的构象调整,这导致了与PAM序列相互作用的蛋白裂隙经历从开放状态到关闭状态的构象变化;同时,Cas12i1的Helical-I结构域、RuvC结构域上的helix-loop基序(helix-loop motif, also termed lid motif)也发生了显著的构象重排,使得它们不再遮挡RuvC结构域上的DNase活性位点,从而释放了Cas12i1对于DNA底物的DNase活性(图4)。研究还表明Cas12i1对DNA底物的切割具有温度和金属离子依赖性。最后,论文讨论了CRISPR-Cas12家族成员Cas12a、Cas12b、Cas12e和Cas12i的结构差异,比较与分析了促使Cas12家族成员形成R-loop结构的关键蛋白基序;并且通过对Cas12i1复合物的结构和生化分析,本项研究给出了一个多步模型来阐述Cas12i1被靶DNA激活及其切割DNA底物的机理。
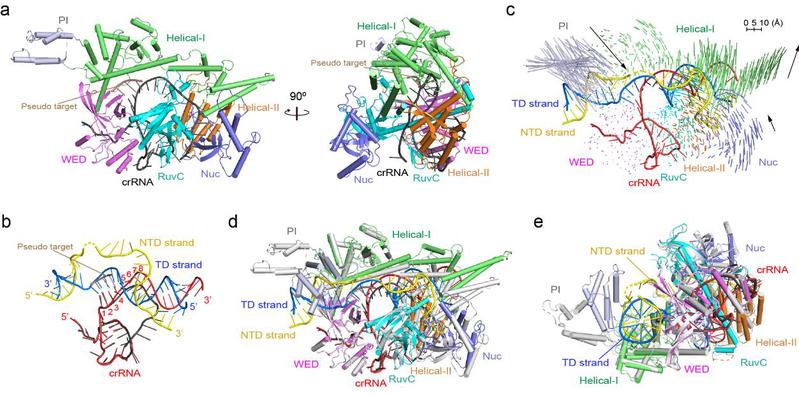
图4. Cas12i1的二元及三元复合物的构象变化
福建师范大学为本研究的唯一完成单位,欧阳松应教授为本文的独立通讯作者,生命科学学院副研究员张博和南方生物医学研究中心研究生罗迪茵、李煜为本文的共同第一作者,该研究得到国家自然科学基金委、福建省科技厅、教育厅、财政厅和福建师范大学等的经费资助,上海同步辐射光源(SSRF)BL-17U1对该研究提供了重要的技术支持。
原文链接:
https://www.nature.com/articles/s41467-021-23876-5