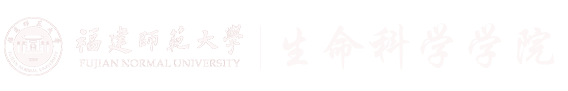CRISPR-Cas系统第二大类成员Cas9效应蛋白在生物学及医学领域具有巨大的应用价值及前景,然而Cas9在切割外源核酸序列时受到特定PAM序列的限制及存在可能的脱靶问题。因此,研究人员从未间断寻找新型的第二大类Cas效应蛋白来扩充基因编辑工具箱中可供选择的工具种类。2019年1月,美国Arbor生物技术公司对不断增加的原核生物基因组及宏基因组数据进行生物信息学挖掘,鉴定出与已知第二大类Cas效应蛋白存在明显差异的Cas12c效应蛋白。Cas12c切割双链靶DNA时,其靶DNA的PAM序列比第二大类Cas效应蛋白SpCas9、Cas12a及Cas12b的PAM序列的限制性都小,这个特征极大的拓展了Cas12c发挥基因编辑功能的靶标空间。然而,Cas12c如何识别single guide RNA(简称sgRNA)、结合并切割靶DNA,以及Cas12c如何识别限制性小的PAM序列,对应的结构基础与分子机理并不清楚。

2022年11月2日,《Nucleic Acids Research》杂志在线发表了福建师范大学欧阳松应教授课题组关于CRISPR-Cas系统第二大类效应蛋白Cas12c1的最新研究成果,论文标题为“Structural insights into target DNA recognition and cleavage by the CRISPR-Cas12c1 system”。该研究报道了Cas12c1效应蛋白与sgRNA(通过融合crRNA与tracrRNA获得)及含有5′-TG-3′PAM的双链靶DNA形成的三元复合物的晶体结构;通过结构分析与生化实验,揭示了Cas12c1识别sgRNA、PAM序列的结构基础,以及Cas12c1通过关键氨基酸残基与蛋白基序参与双链靶DNA的识别、解旋及切割的分子机理;该研究结果为基因编辑工作中有效使用CRISPR-Cas12c1系统提供了理论指导。这是该课题组继CRISPR-Cas12i1(详见BioArt报道:Nat Comm | 欧阳松应组发表CRISPR-Cas12i效应蛋白结构与功能最新研究成果)、CRISPR-Cas13b(详见BioArt报道:Cell Research | 欧阳松应组在CRISPR-Cas13b结构与功能方面取得重要进展)、CRISPR-Cas13d(详见BioArt报道:Nat Comm | 欧阳松应课题组发表CRISPR-Cas13d最新研究成果)的结构与功能研究之后的又一研究成果。此外,该课题组还报道了相关综述(详见BioArt报道:Adv Sci综述 | 欧阳松应团队总结细菌CRISPR-Cas13系统效应蛋白结构与功能机制、应用及研究前景)。
在本研究报道的三元复合物结构中,sgRNA整体呈倒“H”构象,它与位于其上的Cas12c1的多个结构域结合;靶DNA双链中的靶标DNA链与非靶标DNA链解旋,靶标DNA链与sgRNA的向导序列(guide region)结合并形成14-bp的DNA-RNA异源双链结构(heteroduplex)(图1)。位于Cas12c1的PI结构域的一段柔性loop区域(PI loop)直接参与PAM序列的识别,研究还发现其PI结构域的残基R152及其Rec1结构域的残基N299和R374对于正确识别PAM序列至关重要;Cas12c1可以引起PAM序列内部的构象发生形变,使得PAM序列中的dG(-1*)与dA(-2)之间形成特殊的内部氢键识别(图2)。另外,Rec1结构域的残基N297对于靶DNA双链在PAM序列近端解旋中发挥关键作用。
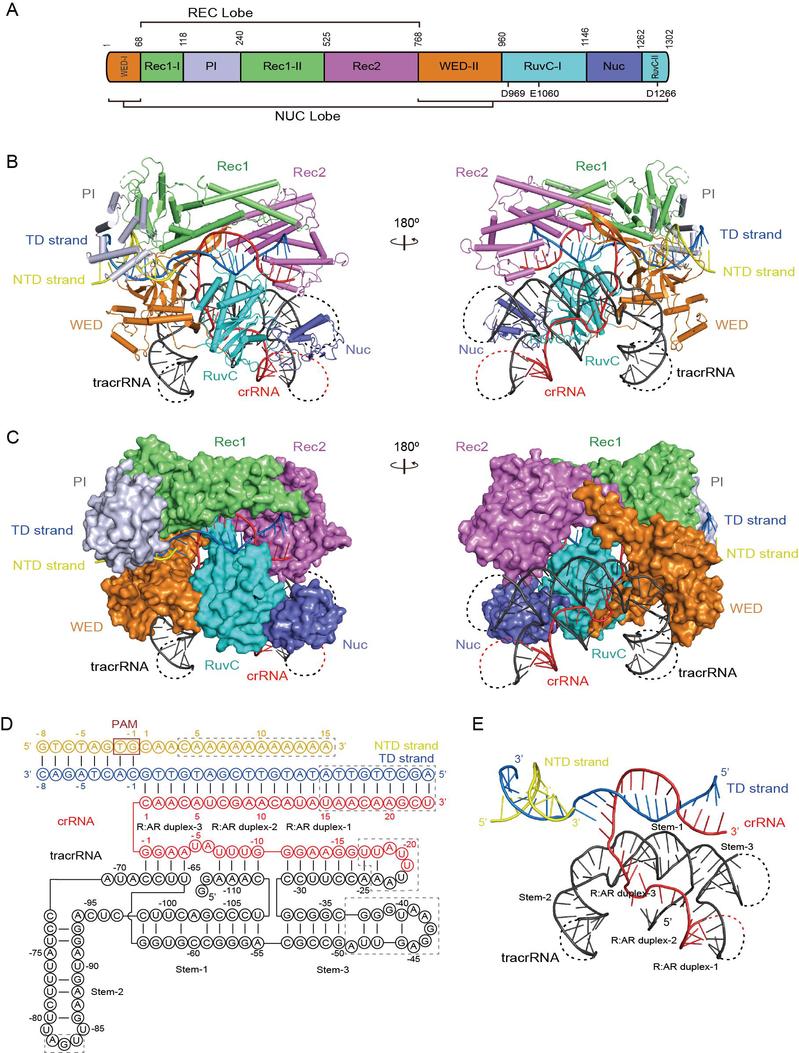
图1. Cas12c1-sgRNA-target DNA三元复合物的晶体结构
研究发现位于sgRNA向导序列的种子区域(seed region)位于其向导序列的前7个核苷酸内,该区域任何一个核苷酸序列的错配将极大地破坏对靶DNA的切割(图3)。需要进一步指出,位于Rec1结构域的螺旋α8在稳定与sgRNA种子区域配对的靶标DNA链的构象中起着重要作用。在Cas12c1的RuvC结构域中,存在两个蛋白基序(即HLH基序、Lid基序),它们彼此相邻并通过一组疏水相互作用保持接触,表明这两个基序在RuvC结构域的DNase活性被激活的构象变化过程中可能存在耦合运动(图3)。此外,RuvC结构域与Nuc结构域表面形成了局部带正电荷的沟槽,这为非靶标DNA链结合到RuvC结构域的DNase活性中心提供可能的路径。
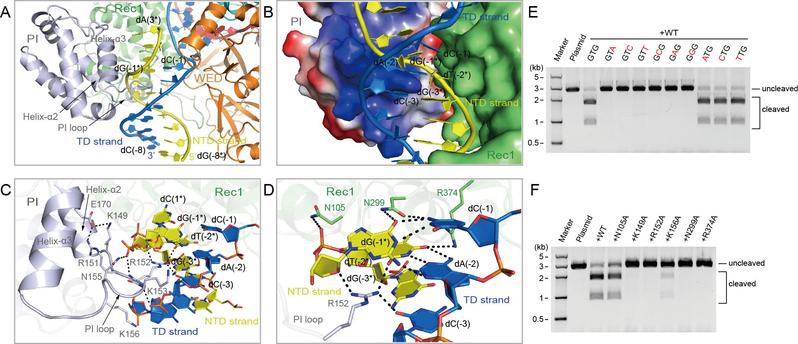
图2. Cas12c1识别PAM序列的结构基础
进一步的体外酶切实验表明,Cas12c1在PAM序列下游第14个核苷酸位置处切割非靶标DNA链,并在PAM序列下游第23个核苷酸位置处切割靶标DNA链(图4)。值得注意的是,Cas12c1在切割靶DNA的两条链时产生相对单一且均一的切割位点,这与第二大类V型CRISPR-Cas家族成员Cas12a、Cas12b、Cas12e、Cas12i和Cas12j在切割靶DNA的两条链时产生多个切割位点相比,Cas12c1作为基因编辑工具可能具有更高的保真性。另外,研究发现Cas12c1以相似的能力切割靶DNA的靶标DNA链与非靶标DNA链,并没有观察到在其他第二大类V型CRISPR-Cas家族成员Cas12a和Cas12i中观察到的Cas效应蛋白对非靶标DNA链比靶标DNA链具有更高切割能力的现象。这可能是由于Cas12c1比其他V型CRISPR-Cas家族成员(比如Cas12a和Cas12i)可以更容易或更早的解旋PAM序列远端的异源双链,从而释放出靶标DNA链,使得靶标DNA链与非靶标DNA链在结合RuvC结构域的DNase活性中心时具有相似的机会。此外,研究还表明双链或单链靶核酸序列至少需要与sgRNA向导序列形成16-bp的配对才能充分激活Cas12c1的DNase活性,而且Cas12c1对靶DNA的切割具有温度与金属离子的依赖性。

图3. Cas12c1识别sgRNA与靶DNA形成的异源双链结构
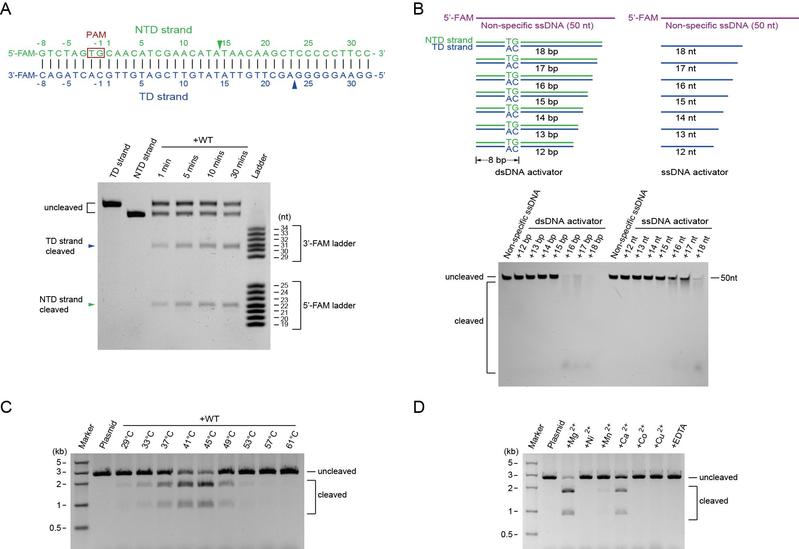
图4. 靶DNA双链的顺式切割及激活Cas12c1的DNA酶切活性的靶DNA长度
在本项研究工作的审稿过程中,东京大学的Nureki课题组报道了Cas12c2与sgRNA及靶DNA的二元、三元复合物冷冻电镜结构研究成果(DOI: 10.1016/j.molcel.2022.03.006)。其研究发现Cas12c2具有加工其前体crRNA(precursor crRNA, 简称pre-crRNA)形成成熟crRNA的能力,可以结合但不具备切割靶DNA的能力,因此该研究缺少与靶DNA切割相关的研究内容。几乎同时,诺贝尔奖得主加州大学伯克利分校的Jennifer A. Doudna课题组在《Molecular Cell》发表研究成果,发现Cas12c2可以加工pre-crRNA,并在sgRNA(由成熟的crRNA与tracrRNA融合改造得到)的引导下,结合而非切割靶基因DNA,从而抑制靶基因的转录并最终发挥抗噬菌体感染的功能(详见BioArt报道:Mol Cell | 天然无DNA核酸酶活性的CRISPR-Cas12c系统可有效抑制基因表达)。与Cas12c2不同,本研究表明Cas12c1不仅可以加工pre-crRNA形成成熟的crRNA,而且可以结合并切割靶DNA。本研究最后分析并讨论了Cas12c1和Cas12c2在识别PAM序列、切割靶DNA存在差异的可能原因,并比较了Cas12c1与其他第二大类V型CRISPR-Cas系统的异同之处。
福建师范大学为本研究的唯一完成单位,生命科学学院、南方生物医学研究中心欧阳松应教授为本文的独立通讯作者,副研究员张博和研究生林锦莹、Vanja Perčulija为本文的共同第一作者,该研究得到国家自然科学基金委、福建省教育厅高水平学科创新平台和福建师范大学等的经费资助,上海同步辐射光源(SSRF)BL-17U1对该研究提供了重要的技术支持。
原文链接:
https://doi.org/10.1093/nar/gkac987