2019年7月22日,我院欧阳松应教授与美国普渡大学罗招庆教授共同通讯在Nature 在线发表了题为“Regulation of phosphoribosyl ubiquitinationby a calmodulin-dependent glutamylase”的研究论文,该研究发现致病性嗜肺军团菌(Legionella pneumophila)效应蛋白SidJ可以被宿主体内的一个钙调蛋白Calmodulin(简称CaM)结合并激活,进而通过谷氨酸化修饰(Glutamylation)抑制SidE家族泛素连接酶活性的分子机理。该工作详细解析了致病性嗜肺军团菌效应蛋白SidJ调控SidE酶活性的分子机制,为探讨谷氨酸修饰酶(glutamylase)的催化机制提供了新的体系,也为临床治疗嗜肺军团菌感染的药物开发提供了新靶点。

嗜肺军团菌是一种引起肺炎的不典型致病菌,其引起的重症肺炎俗称“空调肺”。该菌能通过IV型分泌系统Dot/Icm分泌300多个效应蛋白到宿主细胞中,调控干扰宿主各种信号通路以辅助其自身在宿主细胞内的增殖。对这些效应蛋白的功能解析不仅有助于阐明了嗜肺军团菌侵染宿主的分子机理,同时也能揭示新的细胞信号传导方式和蛋白质翻译后修饰模式【2】。众所周知,泛素化修饰几乎调控真核细胞的生命活动的所有方面3。在与宿主的长期博弈过程中,病原菌进化出了多种方式来干预宿主的泛素修饰系统,从而达到增强侵染效率和逃避宿主免疫监视的目的。2016年普渡大学罗招庆教授课题组在《Nature》杂志上首次报道了SidE家族效应蛋白独立于经典的E1和E2酶的泛素化修饰的重大发现【4】,引发了广泛关注【5-7】,该发现入选为当年度细胞信号传递领域的重大突破之一【8】。后续研究发现嗜肺军团菌中的另一个效应蛋白SidJ可调控SdeA等SidE家族成员【9】,但其生化机制尚未清楚。
针对这一重要科学问题,中美两国研究人员进行了联合攻关。首先,通过质谱(LC-MS/MS)分析,发现SidJ诱导失活的SdeA发生129.01道尔顿的分子量漂移。进一步研究发现,SidJ能对SdeA进行谷氨酸化修饰,且 SdeA 被修饰的位点是位于其mART结构域中860位的谷氨酸残基上,该位点是SdeAmART结构域催化的活性位点之一,主要负责水解NAD+,为泛素激活提供能量。更为有趣的是,研究者通过对SidJ序列分析,发现SidJ的C端具有一个结合CaM的保守IQ 结构域,CaM与SidJ结合形成稳定复合物,进而激活其酶活性(图1)。

![]()
图1 SidJ通过C端I841Q842 motif与CaM结合从而谷氨酸化SdeA
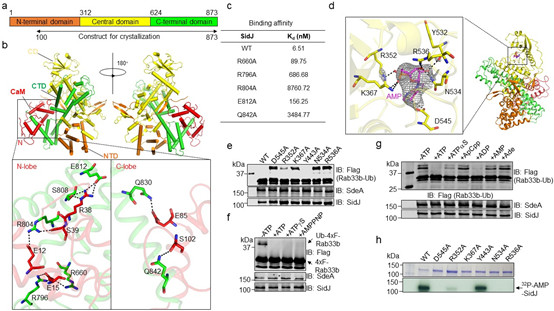
随后研究人员解析了SidJ-CaM二元复合物以及SidJ-CaM结合AMP的三元复合物晶体结构(图2),惊奇地发现SidJ的结构与在已知的真核生物中行使谷氨酸化修饰功能的tyrosine ligase-like(TTLL)家族酶完全不同【10】。结构解析显示CaM能够通过N-lobe和C-lobe与SidJ存在广泛的结合。
![]()
SidJ-CaM(PDB:6K4K)以及SidJ-CaM-AMP(PDB:6K4R)的晶体结构及不同的ATP类似小分子对其激活的能力
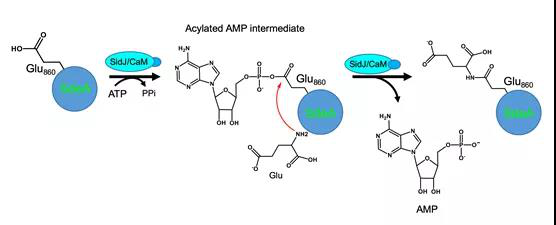
另外,SidJ-CaM 复合体结合一个AMP残基,揭示SidJ是在a位磷酸切割ATP,从而通过酰化单磷酸腺苷化激活被修饰位点的谷氨酸残基,异于常见的磷酸化激活。因此,g 位不能水解的类ATP分子仍能支持其活性。同时,在没有SdeA的反应体系里,SidJ能发生AMP自修饰(self-AMPylation),进一步说明该反应由两步特异的修饰完成(图2)。在没有受体基团的条件下,SidJ将源于ATP的AMP用于自修饰。综合现有的研究结果,研究人员提出了SidJ结合宿主CaM催化SdeA的谷氨酸化修饰,进而导致SdeA失活的分子机制(图3)。
![]()
图3 SidJ-CaM通过形成酰化单磷酸腺苷反应中间物催化谷氨酸化的反应模型。
谷氨酸化修饰此前多见于真核细胞微管蛋白(Tubulin)的修饰,并调控多种重要细胞功能,但是其催化机理如底物识别等机制并不是非常清楚【10,11】。SidJ作为一个CaM依赖的谷氨酸化酶为进一步探讨这一家族酶的作用机制提供了一个窗口。该工作是由美国普渡大学罗招庆教授课题组和福建师范大学欧阳松应教授课题组合作完成的。罗招庆教授和欧阳松应教授为本文的共同通讯作者,普渡大学罗招庆教授课题组博士生干宁海、福建师范大学生命科学学院青年教师甄向凯博士、普渡大学刘尧、以及福建师范大学南方生物医学研究中心一年级硕士生许小龙为共同第一作者。
据悉,《Nature》同期还以背靠背的方式刊登了德国歌德大学(Goethe University)Ivan Dikic教授课题组的研究论文“Inhibition of SidE ubiquitin ligases throughSidJ/Calmodulin catalyzed glutamylation”【12】。美国西南医学中心的研究人员在《Science》上也发表了类似的研究结果【13】。
全文链接:
https://www.nature.com/articles/s41586-019-1439-1
参考文献:
1. Ninghai Gan, Xiangkai Zhen, Yao Liu, Xiaolong Xu, Chunlin He, Jiazhang Qiu,Yancheng Liu, Grant M. Fujimoto, Ernesto S. Nakayasu, Biao Zhou, Lan Zhao, Kedar Puvar, Chittaranjan Das, Songying Ouyang, Zhao-Qing Luo. Regulation of phosphoribosyl ubiquitination by a calmodulin-dependent glutamylase Nature (2019). DOI: 10.1038/s41586-019-1439-1
2. Qiu, J. & Luo, Z. Q. Legionella and Coxiella effectors: strength in diversity and activity. Nat Rev Microbiol 15, 591-605, doi:10.1038/nrmicro.2017.67 (2017).
3. Komander, D. & Rape, M. The ubiquitin code. Annual review of biochemistry 81, 203-229, doi:10.1146/annurev-biochem-060310-170328 (2012).
4. Qiu, J., Sheedlo, M. J., Yu, K., Tan, Y., Nakayasu, E. S., Das, C., Liu, X. & Luo, Z. Q. Ubiquitination independent of E1 and E2 enzymes by bacterial effectors. Nature 533, 120-124, doi:10.1038/nature17657 (2016).
5. Bhogaraju, S. & Dikic, I. Cell biology: Ubiquitination without E1 and E2 enzymes. Nature 533, 43-44, doi:10.1038/nature17888 (2016).
6. Nakasone, M. A. & Huang, D. T. Ubiquitination Accomplished: E1 and E2 Enzymes Were Not Necessary. Mol Cell 62, 807-809, doi:10.1016/j.molcel.2016.06.001 (2016).
7. Du Toit, A. Bacterial pathogenesis: Bacterial effectors skip a few steps. Nat Rev Microbiol 14, 331, doi:10.1038/nrmicro.2016.61 (2016).
8. Adler, E. M. Signaling Breakthroughs of the Year. Sci Signal 9, eg1, doi:10.1126/scisignal.aad9795 (2016).
9. Qiu, J., Yu, K., Fei, X., Liu, Y., Nakayasu, E. S., Piehowski, P. D., Shaw, J. B., Puvar, K., Das, C., Liu, X. & Luo, Z. Q. A unique deubiquitinase that deconjugates phosphoribosyl-linked protein ubiquitination. Cell Res 27, 865-881, doi:10.1038/cr.2017.66 (2017).
10. Rogowski, K., van Dijk, J., Magiera, M. M., Bosc, C., Deloulme, J. C., Bosson, A., Peris, L., Gold, N. D., Lacroix, B., Bosch Grau, M., Bec, N., Larroque, C., Desagher, S., Holzer, M., Andrieux, A., Moutin, M. J. & Janke, C. A family of protein-deglutamylating enzymes associated with neurodegeneration. Cell 143, 564-578, doi:10.1016/j.cell.2010.10.014 (2010).
11. O'Hagan, R., Silva, M., Nguyen, K. C. Q., Zhang, W., Bellotti, S., Ramadan, Y. H., Hall, D. H. & Barr, M. M. Glutamylation Regulates Transport, Specializes Function, and Sculpts the Structure of Cilia. Curr Biol 27, 3430-3441 e3436, doi:10.1016/j.cub.2017.09.066 (2017).
12. Sagar Bhogaraju, F. B., Rukmini Mukherjee, Michael Adams, Moritz M. Pfleiderer, Wojciech P. Galej, Vigor Matkovic, Sissy Kalayil, Donghyuk Shin, Ivan Dikic,. Inhibition of SidE ubiquitin ligases through SidJ/Calmodulin catalyzed glutamylation Nature (2019). DOI:10.1038/s41586-019-1440-8
13. Black, M. H., Osinski, A., Gradowski, M., Servage, K. A., Pawlowski, K., Tomchick, D. R. & Tagliabracci, V. S. Bacterial pseudokinase catalyzes protein polyglutamylation to inhibit the SidE-family ubiquitin ligases. Science 364, 787-792, doi:10.1126/science.aaw7446 (2019).
(文章转自inature公众号)